Reasoning-based drug repurposing
问题&回答
问题
- LLM在Repurposing中的应用情况
- 是否有基于文本结构,引入大模型知识作为一部分特征?
- 测试集是什么
- 传统模型/基于网络的模型的准确率
回答
- 有两种情况。第一种,引入LLM后的文本信息作为向量嵌入到GNN中;第二种,LLM作为筛选数据的方法,从数据层面提升最终效果。
- 有的。将文本以特定的形式组织,如"药物A[SEP]疾病B",然后将其变成向量,嵌入到GNN中。
- 阅读了几篇,测试集有
- B-dataset(SCMFDD-S/L):来自于 Predicting drug-disease associations by using similarity constrained matrix factorization | BMC Bioinformatics | Full Text,这篇文章里主要介绍了一个相似性约束矩阵分解的方法,里面编制了 SCMFDD-S 和 SCMFDD-L 数据集,其中的前者是这里的 B-dataset,适用于常规场景。
- C-dataset:来自于 Drug repositioning based on comprehensive similarity measures and Bi-Random walk algorithm | Bioinformatics | Oxford Academic,由 Dndataset 和 F-dataset 整合而成,结合了药物的 ATC 编码和疾病的 DO 术语。包含 963 种药物、1263 种疾病和 54921 个药物-疾病关系。适用于多源数据融合场景。
- F-dataset:来自于 PREDICT: a method for inferring novel drug indications with application to personalized medicine | Molecular Systems Biology,侧重于模型对文本知识的分析能力。
- R-dataset:整合 C-dataset、F-dataset 和 KEGG 数据库的信息,用于极端稀疏和不平衡数据下的鲁棒性。
- 模型准确率在不同的数据集上不同。这里的B、C、F数据集使用得比较多,一般来说衡量标准是AUC、AUPR、F1-score和Precision。
Action
Week
week1
相关论文调研: 是否有用大模型推理在repurposing里做的? 是否有基于文本&结构特征做repurposing,然后把大模型reasoning的知识作为一部分feature加入,能够提升其效果,找出可复现的工作。
week2
找出可复现的工作(上周遗留), repurposing任务的测试集是什么? 在这个测试集上 传统大模型是多少准确率,基于网络的方式是多少准确率?
相关论文调研
LLM-DDA
问题与回答
Q1. LLM在Repurposing中的应用情况
LLM的推理信息在这里被引入了GNN中作为一部分特征
Q2. 是否有基于文本结构,引入大模型知识作为一部分特征?
文本结构经过LLM编码后的向量,引入到GNN网络中
Q3. 测试集是什么
见下面的数据集部分。一共使用了四个数据集。
Q4. 传统模型/基于网络的模型的准确率
- 与下面四种baseline进行比较
- 机器学习方法:DDA-SKF、NIMCGCN;
- 矩阵分解方法:SCPMF、DRWBNCF;
- 深度学习/GNN方法:REDDA、LAGCN、HDGAT
- LLM 直接预测DirectPred
这里的前三种就是传统模型等其他方法。在下方的结果处说明了AUC、AUPR、F1-score和Precision的值。
解决的问题
现有图神经网络(GNN)方法过度依赖网络拓扑结构,受限于不完整、含噪声的网络数据,且忽略生物医学领域丰富知识的问题。通过整合大语言模型(LLM)推断的知识表示,提升 DDA 预测的准确性和可靠性。
面临挑战
- 数据层面:传统GNN方法以来药物-疾病异构网络存在稀疏性和标签不平衡的问题
- 模型层面:现有方法难以捕捉复杂关联
- 知识利用:LLM生成的离散文本如何转化为适合GNN推理的连续数值表示,并设计高效融合架构
核心方法
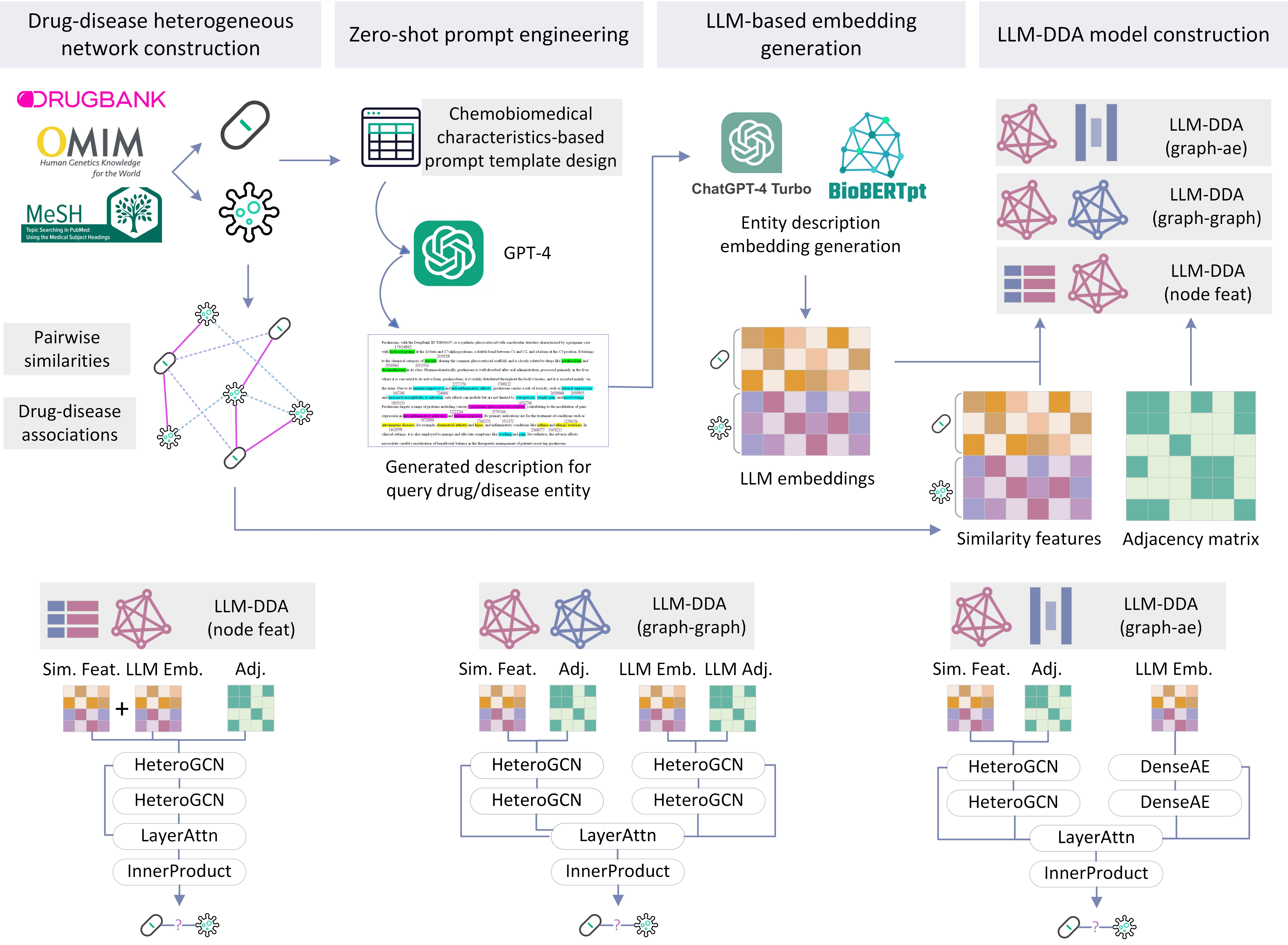
TL;DR:关键在于知识挖掘(LLM 提示)→ 语义编码(嵌入生成)→ 图模型融合(关联预测)
Step1:构建异质网络
Step2:利用LLM的知识来拓展潜在知识
Step3:基于LLM的嵌入生成
Step4:构建LLM-DDA模型
一、药物-疾病异质网络构建(Drug-disease heterogeneous network construction)
- 数据来源:整合 DrugBank(药物数据库 )、OMIM(人类孟德尔遗传数据库 )、MeSH(医学主题词表 )等生物医学数据库 。
- 构建逻辑:
- 先计算 药物-药物、疾病-疾病的成对相似度(Pairwise similarities)(如化学结构、基因功能相似性 )。
- 再结合已知 药物-疾病关联(Drug-disease associations) ,构建成包含药物、疾病节点,以及相似度、关联关系边的异质图网络 ,为后续分析提供拓扑结构基础。
二、零样本提示工程(Zero-shot prompt engineering)
- 核心动作:设计 基于化学生物特征的提示模板(Chemobiomedical characteristics-based prompt template design) ,把药物 / 疾病的专业属性(如靶点、作用通路、临床特征等 )转化为大语言模型(LLM)可理解的指令。
- 功能价值:借助 GPT-4 的知识推理能力,生成目标药物 / 疾病的详细描述 ,挖掘数据库外的潜在知识关联,引入LLM知识。
三、基于 LLM 的嵌入生成
- 双模型进行嵌入生成:
- 用 ChatGPT-4 Turbo(通用大模型)、BioBERTpt(生物医学专用预训练模型 ),对 GPT-4 生成的文本描述做编码。
- 输出 LLM 嵌入,把生物医学语义信息转化为数值向量,让后续图模型能 理解语言知识。
四、LLM-DDA 模型构建(LLM-DDA model construction)
- 多架构覆盖:提供 3 种融合策略,适配不同场景需求:
- LLM-DDA (node feat):直接把 LLM 嵌入作为节点特征,融入传统图神经网络,轻量且易部署。
- LLM-DDA (graph-graph):双图网络并行,分别处理 LLM 嵌入图与原始异质图,再融合结果,强化多源信息互补。
- LLM-DDA (graph-ae):结合图自动编码器(Graph-AE),优化嵌入特征与拓扑结构的融合,适合挖掘深层关联。
- 输入衔接:模型输入关联两部分:
- Similarity features(相似度特征 ):来自第一步异质网络的拓扑计算。
- Adjacency matrix(邻接矩阵):刻画药物-疾病的关联结构,让模型同时学习知识语义(LLM 嵌入)和网络拓扑(图结构),提升药物-疾病关联预测精度。
数据集
四个药物-疾病的基准数据集
| Dataset | Drugs | Diseases | Drug-disease Associations | Pos-Neg Ratio |
|---|---|---|---|---|
| B-dataset | 269 | 598 | 18,416 | 11.45% |
| C-dataset | 663 | 409 | 2,532 | 1.57% |
| F-dataset | 593 | 313 | 1,933 | 1.05% |
| R-dataset | 894 | 454 | 2,704 | 0.67% |
- B-dataset:来自于Predicting drug-disease associations by using similarity constrained matrix factorization | BMC Bioinformatics | Full Text,这篇文章里主要介绍了一个相似性约束矩阵分解的方法,里面编制了SCMFDD-S和SCMFDD-L数据集,其中的前者是这里的B-dataset,适用于常规场景。
- C-dataset:来自于Drug repositioning based on comprehensive similarity measures and Bi-Random walk algorithm | Bioinformatics | Oxford Academic,由Dndataset和F-dataset整合而成,结合了药物的ATC编码和疾病的DO术语。适用于多源数据融合场景。
- F-dataset:来自于PREDICT: a method for inferring novel drug indications with application to personalized medicine | Molecular Systems Biology,侧重于模型对文本知识的分析能力。
- R-dataset:本文自己提出。整合C-dataset、F-dataset和KEGG数据库的信息,用于极端稀疏和不平衡数据下的鲁棒性。
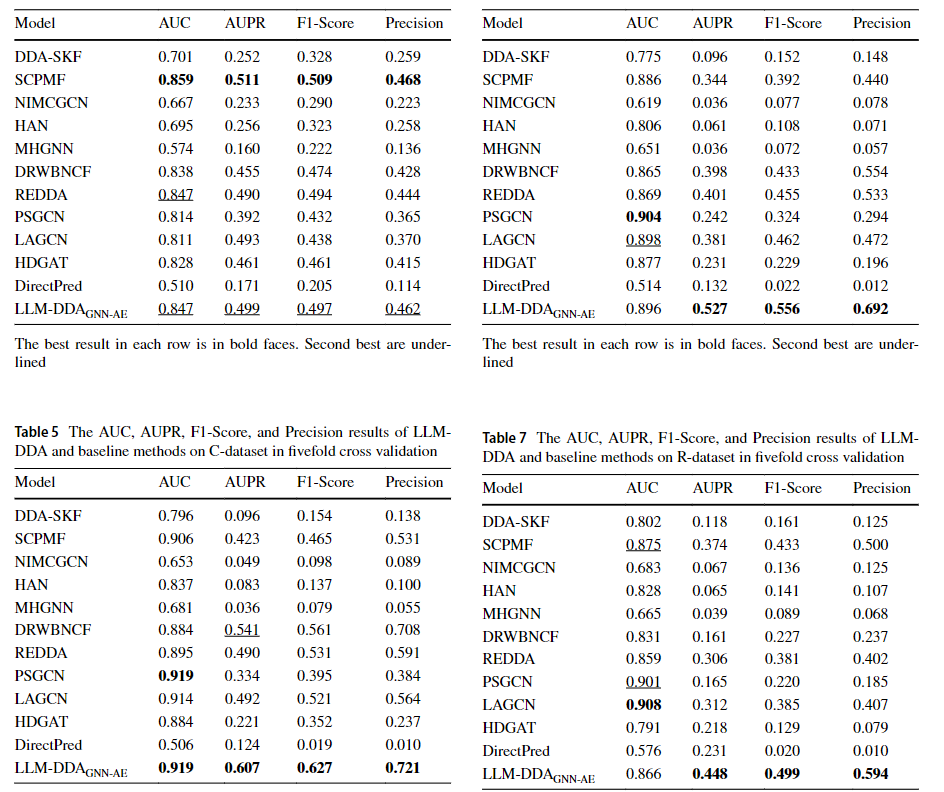
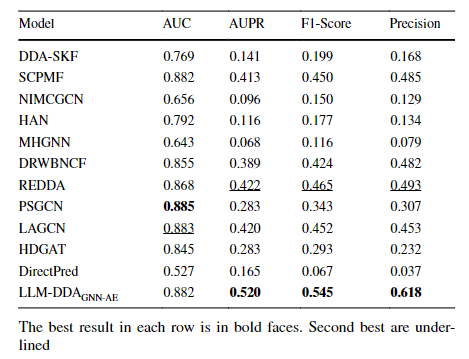
指标结果
- AUC、AUPR、F1和五折交叉验证结果
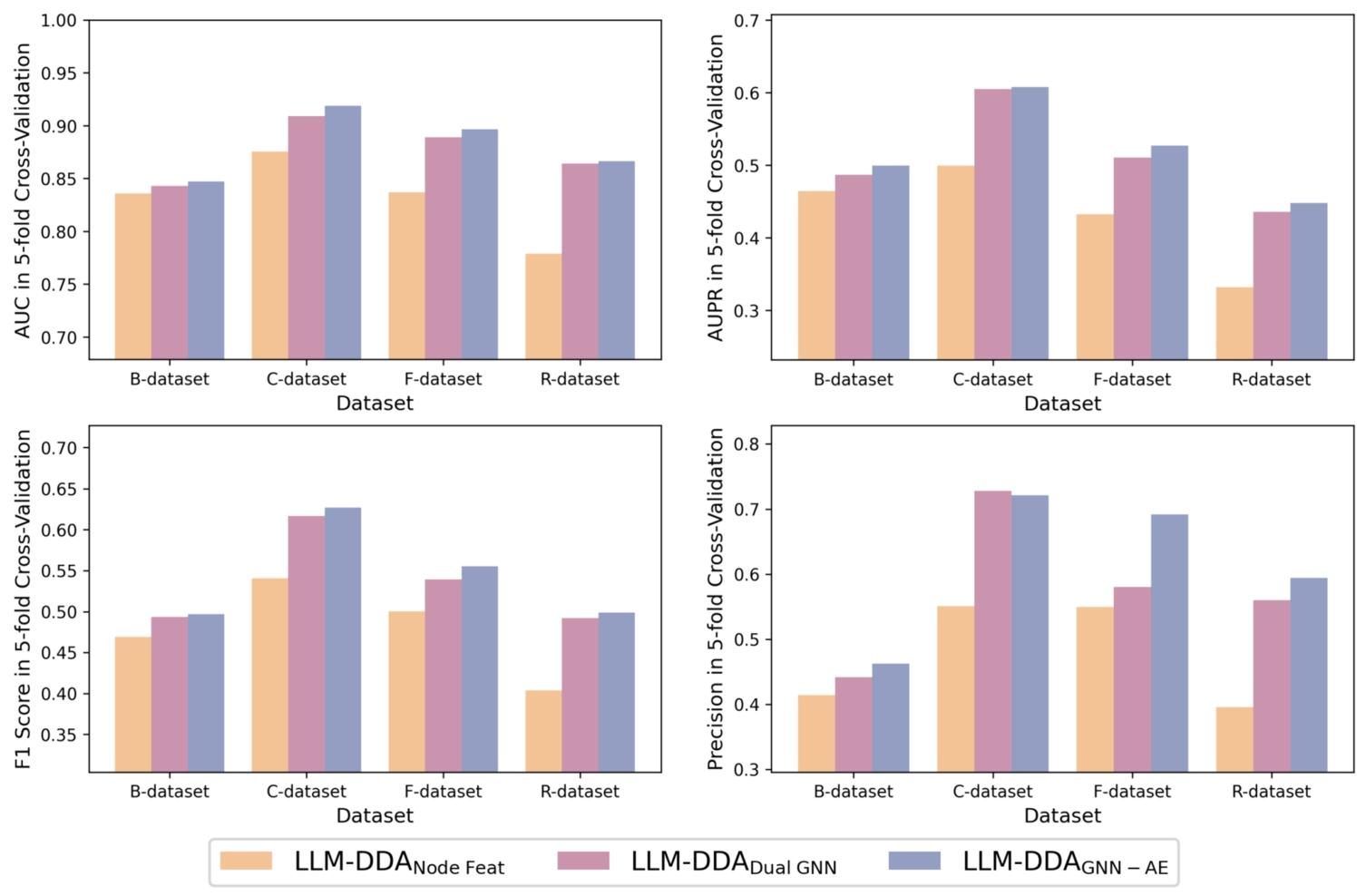
- 与下面四种baseline进行比较
- 机器学习方法:DDA-SKF、NIMCGCN;
- 矩阵分解方法:SCPMF、DRWBNCF;
- 深度学习/GNN方法:REDDA、LAGCN、HDGAT
- LLM 直接预测DirectPred
分别通过AUC、AUPR、F1-score和Precision来判断效果。
LBMFF
Frontiers | Drug–disease association prediction with literature based multi-feature fusion
问题与回答
Q1. LLM在Repurposing中的应用情况
这里使用了BERT来挖掘文献中的关联关系。
Q2. 是否有基于文本结构,引入大模型知识作为一部分特征?
BERT经过预训练和微调之后,能够学习文献中的一些关联关系,输出包含语义关联信息的向量表示,从而文献中的药物-药物相似性关系和疾病-疾病相似性关系知识。
Q3. 测试集是什么
见下面的数据集部分。一共使用了两个数据集。
Q4. 传统模型/基于网络的模型的准确率
对比的模型分为如下部分,都是各自领域的SOTA模型。
- 图神经网络(GNN):DRHGCN、LAGCN、REDDA
- 矩阵分解与正则化:BNNR、DRWBNCF
- 矩阵补全与神经归纳:NIMCGCN
- 核融合与传统机器学习:DDA-SKF
对比结果如研究结果部分所示。
解决的问题
药物重定位中的关联预测问题
面临的挑战
- 文献信息利用不够充分:现在大多数计算方法都是依赖结构化数据库,但是科学文献中有很多未被充分挖掘的药物-疾病关联关系。
- 多特征难以有效融合:药物和疾病的特征具有异质性。
核心方法
提出了LBMFF(基于文献的多特征融合方法)。
- 多源融合:同时用结构化数据库(Drugbank、SIDER 等)和非结构化文献(BERT 处理),挖掘更全面的药物-疾病关联。
- 图网络 + 注意力:用 GCN 处理 “药物-疾病” 关联的图结构,注意力机制强化关键特征,提升预测准确性。
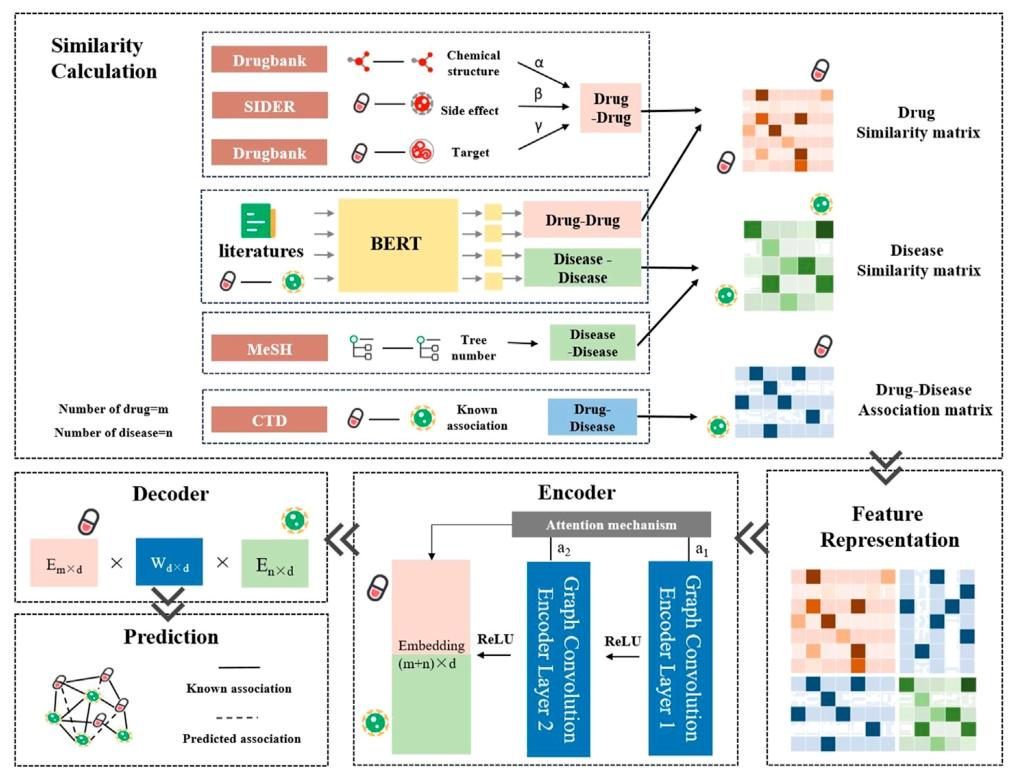
- 相似性计算
- 从多源数据提取特征,计算药物、疾病的相似性,构建关联矩阵
- 药物相似性(Drug-Drug):
整合 3 类结构化数据 + 文献语义:- 化学结构(Drugbank 数据库,药物分子结构)
- 副作用(SIDER 数据库,药物-副作用关联)
- 靶点(Drugbank 数据库,药物-靶点关联)
- 文献语义(BERT 模型处理文献,挖掘药物间语义关联)
最终加权融合(α、β、γ 为权重),输出药物相似性矩阵。
- 疾病相似性(Disease-Disease):
整合 2 类数据: -MeSH 数据库(疾病树编号,疾病分类体系)- 文献语义(BERT 模型处理文献,挖掘疾病间语义关联)
输出疾病相似性矩阵。
- 文献语义(BERT 模型处理文献,挖掘疾病间语义关联)
- 药物-疾病关联(Drug-Disease):
直接从 CTD 数据库(比较毒理学数据库)获取已知关联矩阵,标记已验证的药物-疾病对。
- 特征表示
- 将上一步得到的 药物相似性矩阵、疾病相似性矩阵、已知关联矩阵 拼接,形成模型输入的融合特征矩阵,统一表征药物和疾病的多源信息。
- 编码器
- 用带注意力机制的图卷积网络(GCN) 学习药物、疾病的嵌入表示:
- 两层 GCN(Graph Convolution Encoder Layer)逐层提取特征,ReLU 激活增加非线性。
- 注意力机制(Attention mechanism)动态分配权重,突出关键特征(比如强关联的药物-疾病对)。
- 最终输出 药物 + 疾病的嵌入向量(维度 (m + n)×d ,m 是药物数,n 是疾病数,d 是嵌入维度 )。
- 解码器
- 通过矩阵乘法还原关联预测:
- 药物嵌入($Eₘₓ×d$ )、权重矩阵($W_d×d$ )、疾病嵌入($Eₙₓ×d $)相乘,重建药物-疾病关联得分。
- 通过矩阵乘法还原关联预测:
- 预测输出潜在关联
- 实线:已知关联(来自 CTD 数据库)。
- 虚线:模型预测的潜在关联(未被验证,但算法判定有高可能性的药物-疾病对 ),可辅助药物重定位研究。
BERT在文中
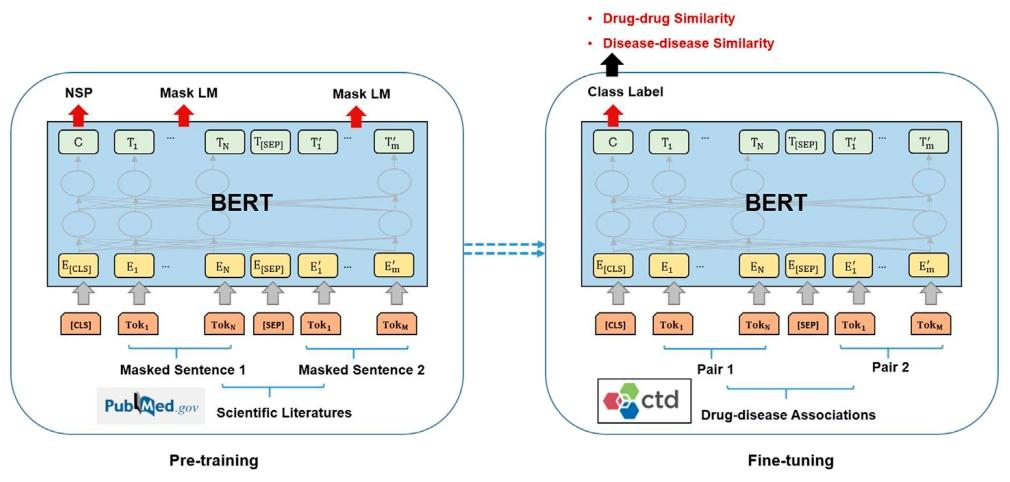
预训练
- 数据准备
- 筛选包含药物、疾病名称的科学文献(如从 PubMed 获取,文中提到的 673,665 篇相关文献 ),这些文献包含药物-疾病关联的语义描述(如治疗、关联、机制等表述 )。
- 将文献中疾病、药物相关文本构建为“药物文本-疾病文本” 对作为输入序列。
- Masked LM
- 让BERT预测被遮盖内容,如[MASK]可以治疗糖尿病,需要推断出是药物X。
- NSP
- 真实对:药物A[SEP]疾病B
- 虚假对:药物C[SEP] 疾病D
- 需要判断文本对是否是真实对
模型微调
- 数据
- 使用CTD等数据库中已知的药物-疾病关联对。存在关联为正例,无关联为负例
- 输出语义特征
- 经过预训练 + 微调后,BERT 对输入的 “药物-疾病文本对”,会输出包含语义关联信息的向量表示
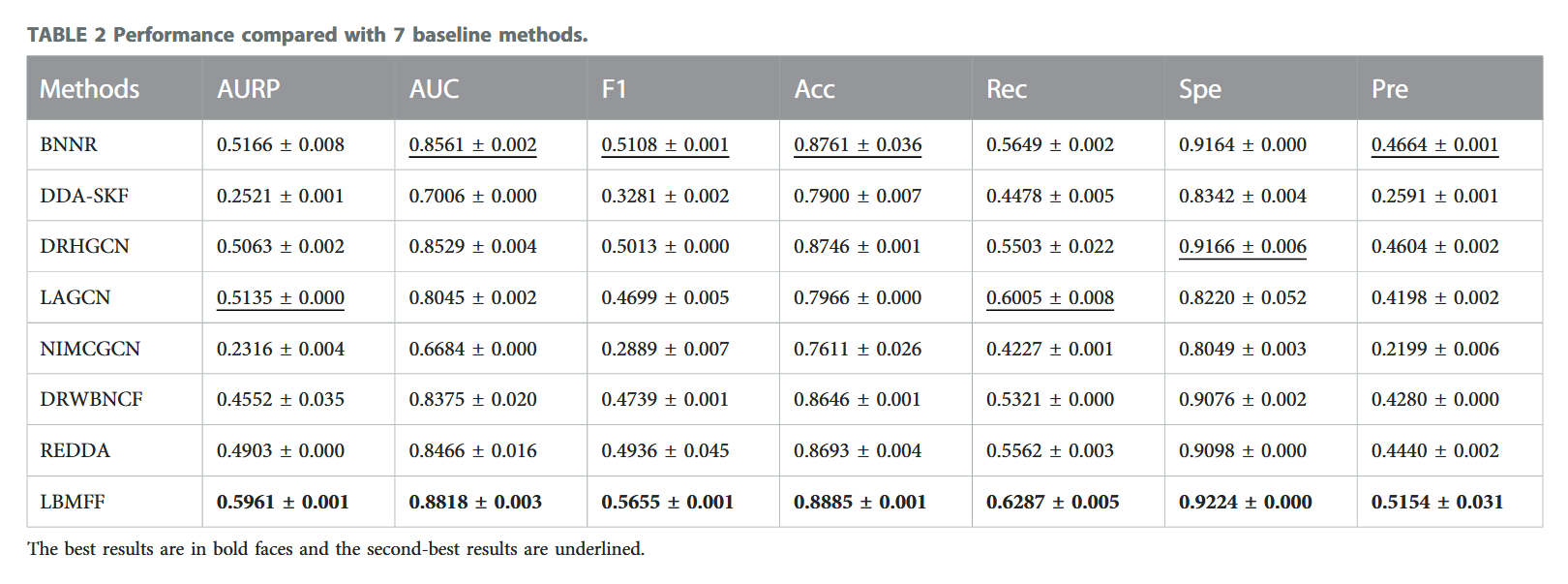
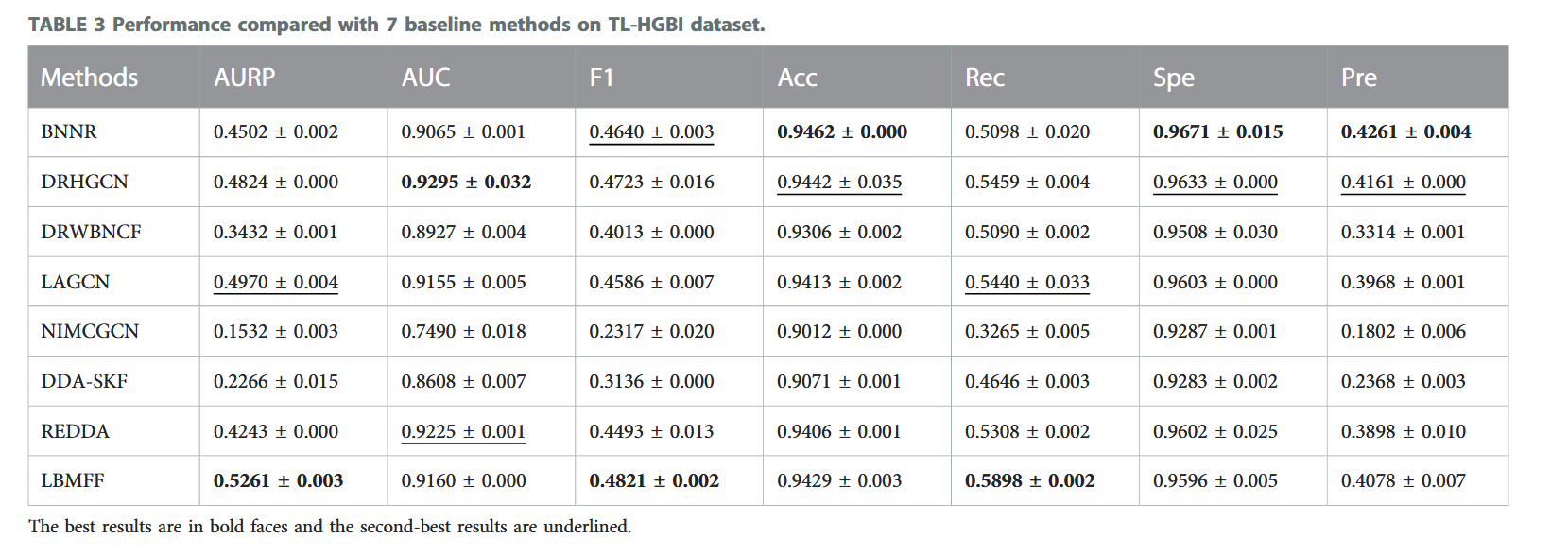
研究结果
测试集
- Zhang(SCMFDD-S):来自于Predicting drug-disease associations by using similarity constrained matrix factorization | BMC Bioinformatics | Full Text,在上面被称为B-dataset。包含269种药物、598种疾病和18416个药物-疾病关系。本文中还从其他数据库中填充了化学结构、药物-靶点关系和药物副作用、疾病树作为药物-药物相似性度量的数据。
- TL-HGBI:来自于Drug repositioning by integrating target information through a heterogeneous network model | Bioinformatics | Oxford Academic。包含963种药物、1263种疾病和54921个药物-疾病关系。同样地,也进行了信息填充来进行相似性度量。
指标表现
和SOTA模型进行比较,分别是
- 图神经网络(GNN):DRHGCN、LAGCN、REDDA
- 矩阵分解与正则化:BNNR、DRWBNCF
- 矩阵补全与神经归纳:NIMCGCN
- 核融合与传统机器学习:DDA-SKF
LBMFF在所有指标上都体现出了领先。
此外,LBMFF在TL-HGBI在多数指标上仍优于对比方法,证明方法在大规模数据集上的泛化能力。
LLM进行阴性数据标注
Improving drug repositioning with negative data labeling using large language models-PubMed
问题与回答
Q1. LLM在Repurposing中的应用情况
LLM引入了自己的推理能力来修改训练数据。
Q2. 是否有基于文本结构,引入大模型知识作为一部分特征?
没有。这里大模型的作用是通过自身的知识,在文本中筛选出更好的数据来提升最终效果的。
Q3. 测试集是什么
测试集是由AACT 数据库得到。作者手动选择了一部分数据(5 阳性 + 11 阴性 )作为测试集验证泛化能力。
Q4. 传统模型/基于网络的模型的准确率
见研究结果。本文通过提升数据集中真阴性数据的质量来提高了最终的模型效果。具体来说,提升的效果还是比较显著的。
解决的问题
在药物重新定位中,监督机器学习模型因缺乏可靠的阴性数据(即因无效或毒性失败的药物)而预测准确性和泛化能力不足。
传统的 Positive-Unlabeled(PU)学习方法通过随机采样或简单分类未标记数据作为阴性,存在误分类或决策边界简化的问题,导致模型性能受限。
LLM通过进行分析文献和数据,系统挖掘真阴性数据,提升了阴性数据的质量。
面临的挑战
- 阴性数据获取困难:多数数据库会把试验中止就等同于阴性,但终止原因可能并不是因为药物疗效/毒性问题,而是资金不足等原因。
- PU学习方法的局限性:随机采样未标记数据作为阴性会引入分类偏差,而基于聚类等策略的阴性筛选假设正负样本边界清晰,与真实场景不符,导致模型泛化能力差。
- 临床数据处理复杂性:临床试验文本存在表述不规范、结果不完整等问题,传统方法难以准确解析并识别真正的阴性药物。
核心方法
GPT-4 系统性挖掘真阴性数据,为药物重定位提供 “高质量标注 + 精准模型” 方案,可扩展到其他疾病。
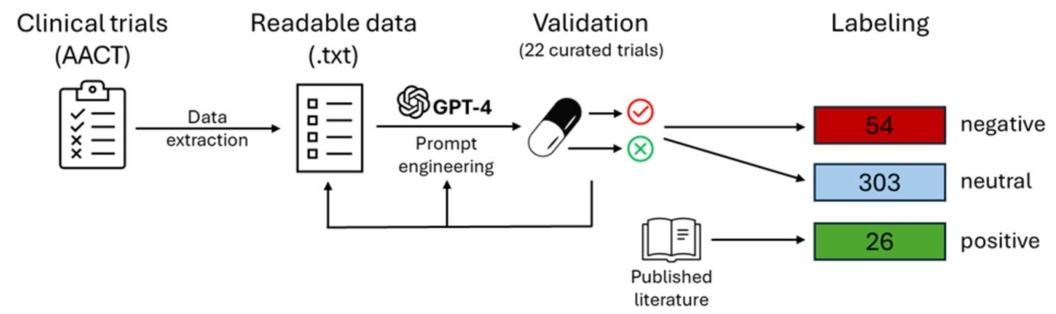
- 临床数据获取:从 AACT 数据库获取 2539 个前列腺癌相关临床试验,排除未公布结果和因非疗效原因终止的试验后,剩余 1442 个试验由 GPT-4 分析。
- GPT-4的训练和验证:使用 22 个手动策划的试验对 GPT-4 进行验证,其中包括 14 个无关试验、4 个阳性试验和 4 个阴性试验,GPT-4 成功正确分类所有试验。
- 特征构建:
- 知识-based 特征:从 DrugBank 获取药物-基因、药物-靶点、药物-通路相互作用、结构特性和靶点类别等,转换为独热编码。
- network-based 特征:构建包含药物-药物相似性、蛋白质-蛋白质相互作用和基因本体论的多层生物网络,提取拓扑特征。
- 对比策略:
- GPT-4 标注:使用 GPT-4 识别的 26 个阳性和 54 个阴性药物。
- 无采样 PU:将所有未标记药物视为阴性,导致类别不平衡。
- 下采样 PU:从未标记药物中随机采样 43 个阴性,重复 100 次以控制类别不平衡。
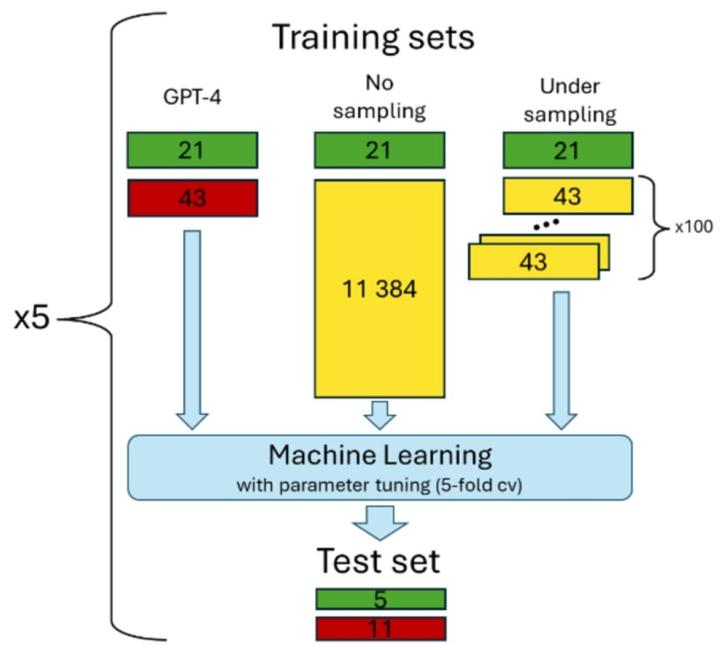
训练集和测试集来自于作者自己收集和整理。
研究结果
使用六种机器学习算法和5折交叉验证训练模型,测试集验证泛化能力(5 阳性 + 11 阴性 )。
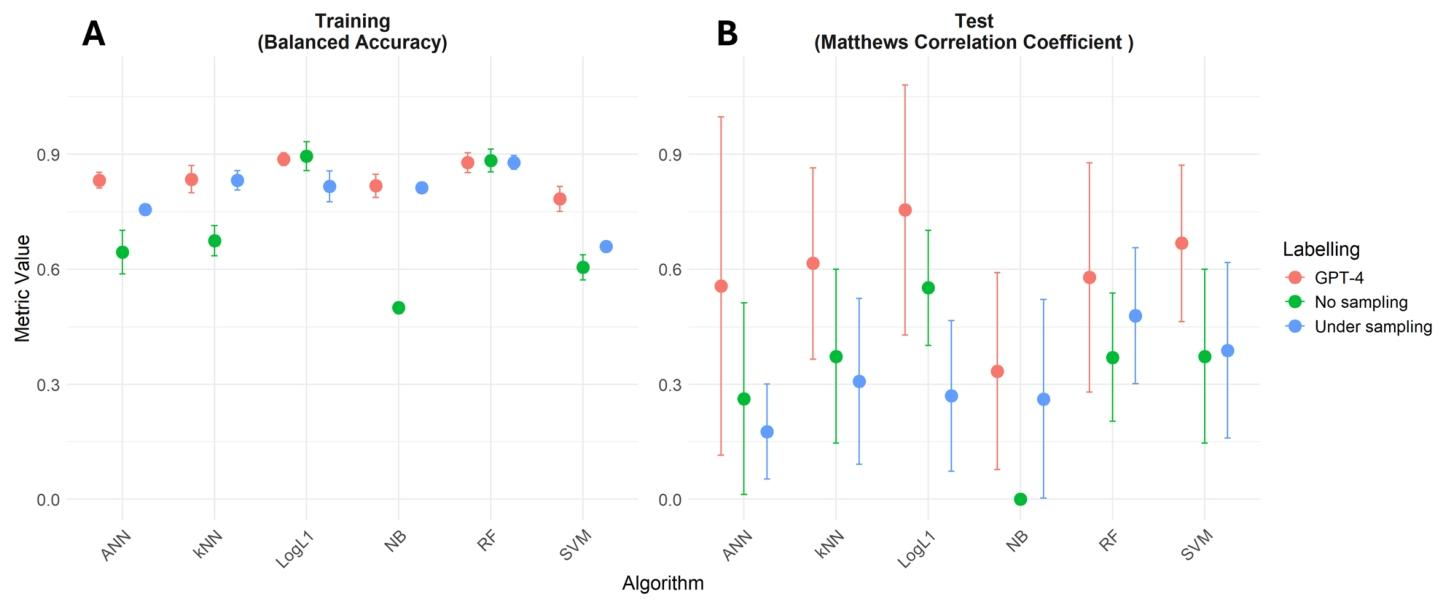
1. 训练集表现(图 A):
- GPT-4(🔴):多数算法(如 LogL1、RF、SVM )平衡准确率接近 0.9,说明用 GPT-4 标注的高质量数据,模型在训练集上 “学的好”,分类稳定。
- No sampling(🟢):部分算法(如 ANN、NB )准确率暴跌,因阴性样本太多,学不到有效模式。
- Under sampling(🔵):表现居中,虽平衡了类别,但随机丢数据,训练效果不如 GPT-4 标注。
2. 测试集表现(图 B):
- GPT-4(🔴):多数算法(如 LogL1、RF、SVM )MCC 显著高于其他策略,尤其是 LogL1、RF,说明模型泛化能力强,用 GPT-4 标注数据训练的模型,在真实测试集上 “预测准”。
- No sampling(🟢):MCC 普遍低(如 NB 甚至接近 0 ),模型被类别不平衡拖垮。
- Under sampling(🔵):MCC 比 GPT-4 低,因下采样丢了很多真实阴性信息,模型学到的规律不完整。
其他
DrugGen
这篇文章介绍了一个基于DrugGPT的模型DrugGen,通过监督微调、近端策略优化和强化学习来引导模型生成更高质量的分子。
DrugReAlign
提出了一个多源提示的LLM药物重定位框架。
Benchmark:LLM在八项化学任务中的表现
What can Large Language Models do in chemistry? A comprehensive benchmark on eight tasks
评估了LLM在八项具体的化学任务中的表现,采用零样本、少样本上下文学习设置,最后得出GPT-4最佳,Davinci-003次之,GPT-3.5第三。
BioBERT
介绍了一个用于生物医学领域的预训练语言表示模型,其在BERT的基础上使用生物医学领域语料库进行预训练。