数据库
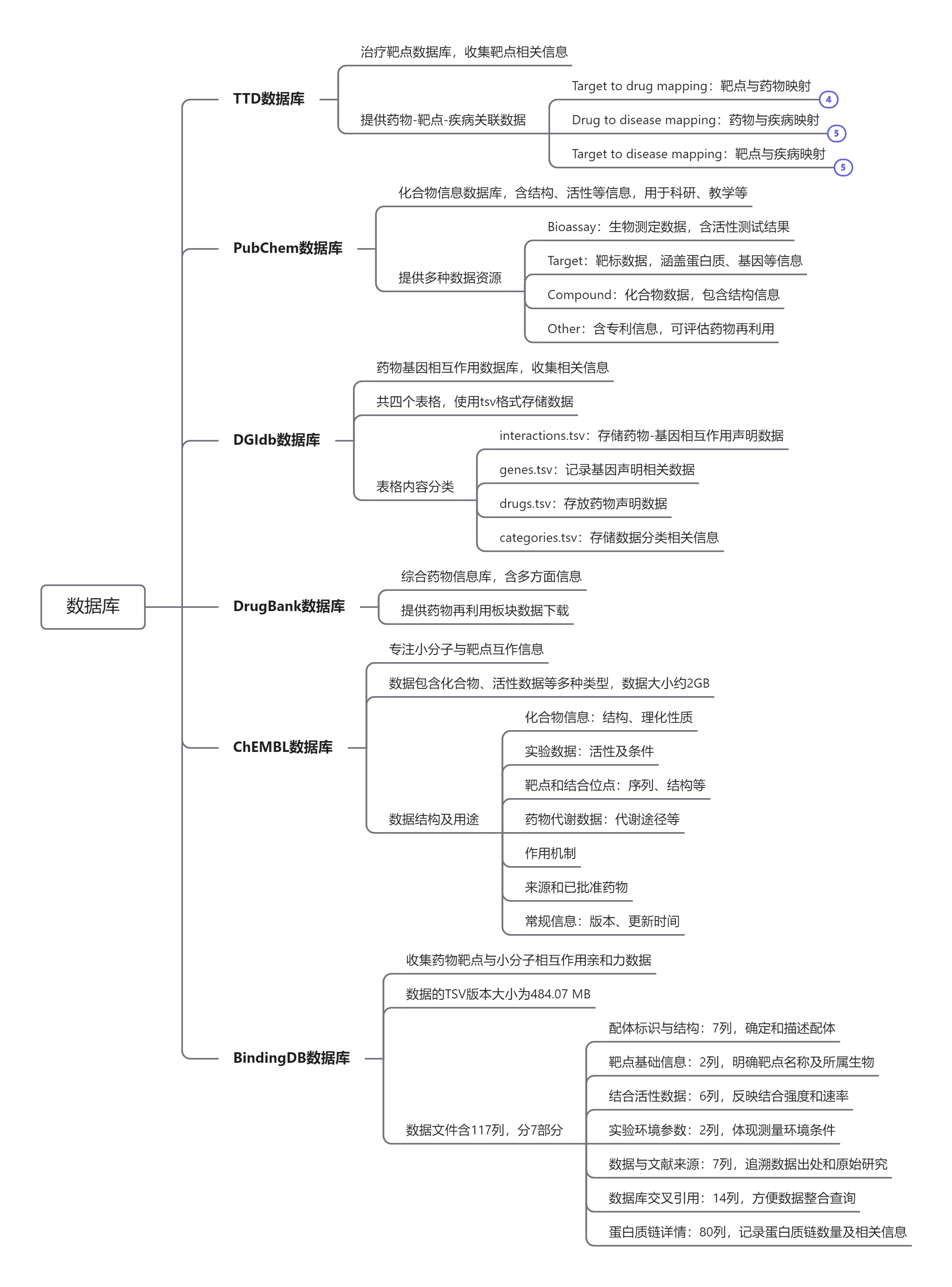
TTD
- TTD
- 治疗靶点数据库。它是一个专门收集和整理与治疗靶点相关信息的数据库。
- 下载地址:Full Data Download | Therapeutic Target Database
- 数据库比较大,这里筛选了药物-靶点-疾病关联数据:
- Target to drug mapping with mode of action
TargetID:靶点的唯一标识符。可以在数据库中找到靶点的详细信息。DrugID:药物的唯一标识符。可以获取该药物的相关信息。Highest_status:表示药物在研发或临床应用中的最高阶段或状态,Approved说明该药物已经通过了相关监管机构的审批,被批准用于临床治疗。MOA:即Mode of Action,作用模式的缩写,Modulator表明该药物对靶点的作用方式是作为调节剂。
- Drug to disease mapping with ICD identifiers
TTDDRUID:表示 TTD 药物 ID,即该药物的唯一标识。DRUGNAME:药物名称。INDICATI:适应症,即药物被用于治疗的病症。Disease entry:疾病条目,这里使用了 ICD-11 编码来表示具体的疾病。Clinical status:临床状态,指药物在临床试验或临床应用中的阶段或情况,如已批准、处于某一阶段的临床试验等。
- Target to disease mapping with ICD identifiers
TARGETID:代表 TTD 靶点 ID,为靶点在数据库内的唯一识别编号。TARGNAME:靶点的具体名称。INDICATI:适应症,即药物被用于治疗的病症。Disease entry:疾病条目,这里使用了 ICD-11 编码来表示具体的疾病。Clinical status:临床状态。
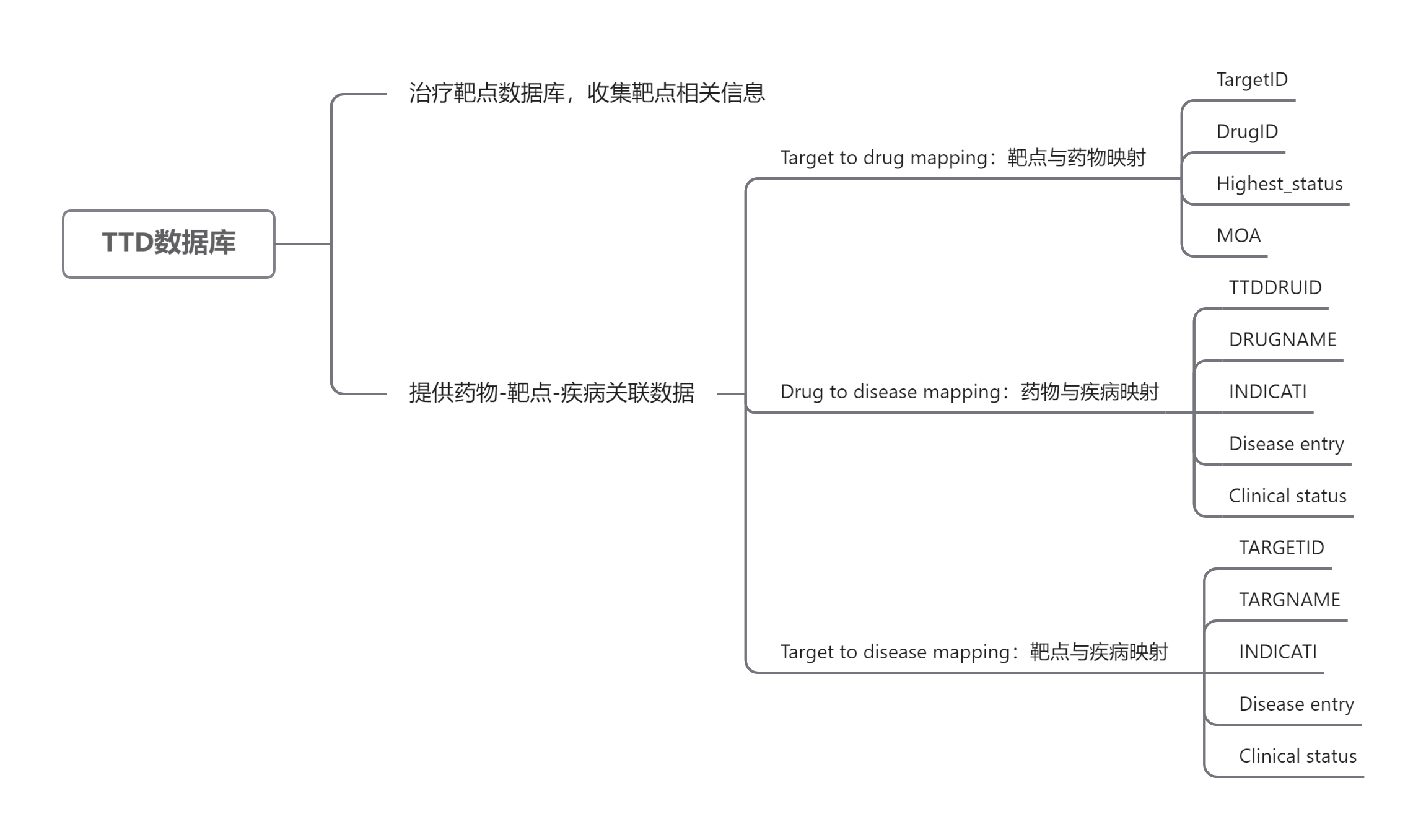
- Target to drug mapping with mode of action
表一:Target to drug mapping with mode of action
| TargetID | DrugID | Highest_status | MOA |
|---|---|---|---|
| T87024 | D00RRU | Approved | Modulator |
这个表格表示:ID为D00RRU的药物能够以调节剂的方式作用ID为T87024的疾病,并且已经被批准。
表二:Drug to disease mapping with ICD identifiers
| TTDDRUID | DRUGNAME | INDICATI | ICD-11 | Clinical status |
|---|---|---|---|---|
| DZB84T | Maralixibat | Pruritus | EC90 | Approved |
| DZB84T | Maralixibat | Progressive familial intrahepatic cholestasis | 5C58.03 | Phase 3 |
| DZB84T | Maralixibat | Alagille syndrome | LB20.0Y | Phase 2 |
这个表格表示ID为DZB84T的药物,名为Maralixibat,有对应几种适应症:
- 适应症为
Pruritus(瘙痒症),ICD-11 编码是EC90,临床状态为Approved(已批准)。 - 适应症为
Progressive familial intrahepatic cholestasis(进行性家族性肝内胆汁淤积症),ICD-11 编码是5C58.03,临床状态为Phase 3(三期临床试验)。 - 适应症为
Alagille syndrome(阿拉基综合征),ICD-11 编码是LB20.0Y,临床状态为Phase 2(二期临床试验)。
表三:Target to disease mapping with ICD identifiers
| TARGETID | TARGNAME | INDICATI | INDICATI | INDICATI |
|---|---|---|---|---|
| T00033 | Transforming growth factor alpha (TGFA) | Phase 1/2 | Chronic kidney disease | [ICD-11: GB61] |
这个表格表示 ID 为 T00033 的靶点,名为 Transforming growth factor alpha (TGFA),有对应的临床关联信息:关联的疾病适应症为Chronic kidney disease(慢性肾脏病),ICD-11 编码是GB61,临床状态为Phase 1/2(一期 / 二期临床试验 )。
PubChem
- PubChem
- PubChem是美国国立卫生研究院的一个开放化学数据库。化合物信息数据库,收录大量化合物的结构、生物活性等信息,为科研、药物研发等提供数据支持,也用于化学知识普及和教学。这个数据库非常大。
- 用途参考:Nucleic Acids Research | 疗效药物靶标的比较性研究与数据平台构建。
- FTP下载:Index of /pubchem
- 药物-疾病关联数据
./Bioassay:生物测定数据,包含大量药物对不同生物靶点或细胞系的活性测试结果./Target:靶标数据,涵盖蛋白质、基因、通路和分类学等信息。./Commpound和./Compound_3D:化合物的数据信息包含结构信息。./Other:GooglePatents和IBM中包含专利信息。寻找专利即将过期/已过期的药物,并对这些药物进行再利用评估。
下面以./Bioassay数据为例。
表 1:PubChem的./Bioassay的CSV文件的标题行的分类和标签说明
| 分类 | 标签名称 | 说明 | |
|---|---|---|---|
| 数据行 | PUBCHEM_RESULT_TAG | 行ID | |
| 数据行 | PUBCHEM_SID | PubChem SID | |
| 数据行 | PUBCHEM_CID | PubChem CID | |
| 数据行 | PUBCHEM_ACTIVITY_OUTCOME | PubChem活性结果(即,Inactive, Active, Inconclusive, Unspecified, or Probe) | |
| 数据行 | PUBCHEM_ACTIVITY_SCORE | PubChem活性得分,值越高表示活性越强 | |
| 数据行 | PUBCHEM_ACTIVITY_URL | 测试结果特定的url | |
| 数据行 | PUBCHEM_ASSAYDATA_COMMENT | 测试结果特定的注释 | |
| 数据行(可选) | 测试结果名称1(如:name of test result 1) | 测试结果1的数据 | |
| 数据行(可选) | 测试结果名称2(如:name of test result 2) | 测试结果2的数据 | |
| 可选标题行(测试结果) | RESULT_UNIT | 单位(e.g. MICROMOLAR, NANOMOLAR和其他PubChem上传系统使用的标签) | |
| 可选标题行(测试结果) | RESULT_IS_ACTIVE_CONCENTRATION | 如果测试结果表示有效浓度则为TRUE | |
| 可选标题行(测试结果) | RESULT_IS_ACTIVE_CONCENTRATION_QUALIFIER | 如果测试结果表示与有效浓度相关的终点限定词(e.g. <, <=, =, >, >=)则为TRUE | |
| 可选标题行(测试结果) | RESULT_ATTR_CONC_MICROMOL | 以微摩尔为单位的测试浓度 | |
表二:PubChem CSV/Data/0000001_0001000/1.csv文件
| PUBCHEM_RESULT_TAG | PUBCHEM_SID | PUBCHEM_CID | PUBCHEM_EXT_DATASOURCE_SMILES | PUBCHEM_ACTIVITY_OUTCOME | PUBCHEM_ACTIVITY_SCORE | PUBCHEM_ACTIVITY_URL | PUBCHEM_ASSAYDATA_COMMENT | LogGI50_M | LogGI50_u | LogGI50_V | IndnGI50 | StddevGI50 | LogTGI_M | LogTGI_u | LogTGI_V | IndnTGI | StddevTGI |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 66954 | 11122 | CC1=CC(=O)C=CC1=O | Inactive | 10 | http://dtp.nci.nih.gov/dtpstandard/servlet/doseresponse?searchtype=NSC&searchlist=1&systemname=NCI+Cancer&idn1=1&idn2=1 | -4.5753 | 1 | 0 | -4 | 1 | 0 |
这个表格表示数据行 ID 为 1 的记录,其中涉及的 PubChem SID 为 66954,PubChem CID 为 11122,化合物的 SMILES 表示形式为CC1=CC(=O)C=CC1=O,有对应的生物活性关联信息:
- PubChem 活性结果为
Inactive(无活性),PubChem 活性得分为 10,表明在此次生物测定中该化合物活性较低。 - 测试结果特定的 url 为dtp.cancer.gov/services/nci60data/colordoseresponse/pdf/1,可通过该链接获取更多相关测试结果信息。
- 测试结果特定的注释为空。
- 关于 GI50(半数生长抑制浓度)的相关数据:
- 以摩尔为单位的 GI50 结果的对数
LogGI50_M为 - 4.5753。 - 测试次数平均数量
IndnGI50为 1。 - 对所有测试的 GI50 结果的对数(Log10)的标准偏差
StddevGI50为 0,说明该测量值较为稳定。
- 以摩尔为单位的 GI50 结果的对数
- 关于 TGI(总生长抑制)的相关数据:
- 以摩尔为单位的 TGI 结果的对数
LogTGI_M为 - 4。 - 测试次数平均数量
IndnTGI为 1。 - 对所有测试的 TGI 结果的对数(Log10)的标准偏差
StddevTGI为 0,表明该测量值稳定性较好 。
- 以摩尔为单位的 TGI 结果的对数
其他的如./Target、./Commpound和./Compound_3D等数据也呈现如此结构。
DGIdb
- DGIdb
- 药物基因相互作用数据库,主要收集和整理药物与基因之间相互作用的相关信息。
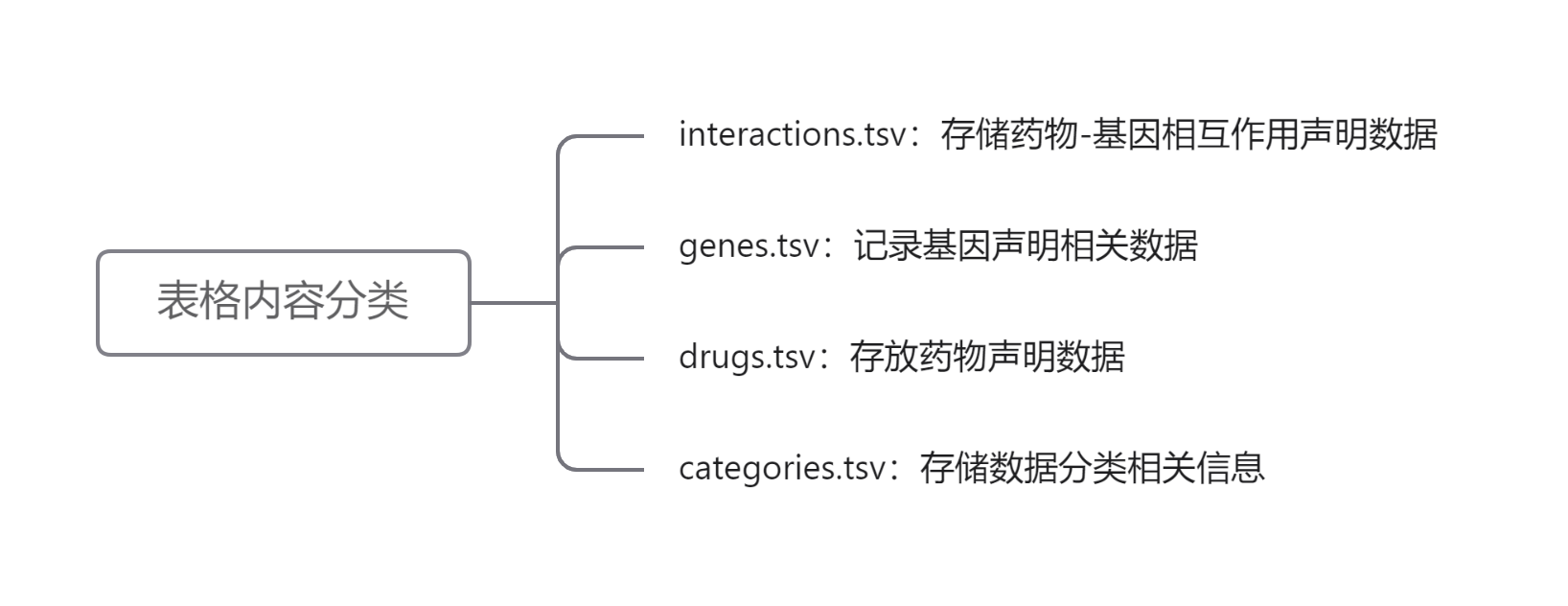
- 这个数据库一共就四个表格,分别存储药物-基因相互作用、基因、药物和分类相关信息,使用
tsv格式进行存储。数据大小约为24.9MB。- interactions.tsv:存储所有药物-基因相互作用声明数据,包含不同药物与基因之间相互作用的信息。
- genes.tsv:记录了基因声明相关数据,可能涵盖基因的基本信息,如基因名称、基因 ID、基因功能注释等内容。
- drugs.tsv:存放药物声明数据,包括药物的名称、药物 ID、药物的基本属性、药理作用等信息。
- categories.tsv:用于存储与数据分类相关的信息。
表一:interactions.tsv
其中的各列的意思分别是
- gene_claim_name:基因的声明名称。
- gene_concept_id:基因的唯一标识代码。
- gene_name:基因的标准正式名。
- interaction_source_db_name:基因与药物相互作用数据的来源数据库名。
- interaction_source_db_version:来源数据库的版本。
- interaction_type:基因和药物间相互作用的类别。
- interaction_score:体现基因与药物相互作用强度的数值。
- drug_claim_name:药物的特定称谓。
- drug_concept_id:药物的唯一标识代码。
- drug_name:药物的标准正式名。
- approved:药物是否已获批上市的标识。
- immunotherapy:药物是否属于免疫治疗药物的标识。
- anti_neoplastic:药物是否具有抗肿瘤作用的标识。
| gene_claim_name | gene_concept_id | gene_name | interaction_source_db_name | interaction_source_db_version | interaction_type | interaction_score | drug_claim_name | drug_concept_id | drug_name | approved | immunotherapy | anti_neoplastic |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CYP2D6 | hgnc:2625 | CYP2D6 | DTC | 9/2/20 | NULL | 0.017709164 | RACLOPRIDE | ncit:C152139 | RACLOPRIDE | FALSE | FALSE | FALSE |
| PPARG | hgnc:9236 | PPARG | DTC | 9/2/20 | NULL | 0.84012274 | KALOPANAX-SAPONIN F | chembl:CHEMBL1833984 | CHEMBL:CHEMBL1833984 | FALSE | FALSE | FALSE |
这个表格有两列,这里只对第一列进行解释:基因声明名称为CYP2D6,基因概念 ID 是hgnc:2625,基因名称同样为CYP2D6。相互作用来源数据库名称是DTC,数据库版本为9/2/20,相互作用类型为空(NULL),相互作用得分为0.017709164。药物声明名称是RACLOPRIDE,药物概念 ID 为ncit:C152139,药物名称是RACLOPRIDE。该药物未被批准(approved为FALSE),不是免疫疗法(immunotherapy为FALSE),也不是抗肿瘤药物(anti_neoplastic为FALSE) 。
表二:drugs.tsv
其中的各列的意思分别是
- drug_claim_name:药物的特定称谓。
- nomenclature:药物命名法类型。
- concept_id:药物的唯一标识代码。
- drug_name:药物的标准正式名。
- approved:药物是否已获批上市的标识。
- immunotherapy:药物是否属于免疫治疗药物的标识。
- anti_neoplastic:药物是否具有抗肿瘤作用的标识。
- source_db_name:药物数据的来源数据库名。
- source_db_version:来源数据库的版本。
| drug_claim_name | nomenclature | concept_id | drug_name | approved | immunotherapy | anti_neoplastic | source_db_name | source_db_version |
|---|---|---|---|---|---|---|---|---|
| BRAF(V600E) Kinase Inhibitor RO5212054 | Primary Drug Name | ncit:C92591 | BRAF(V600E) KINASE INHIBITOR RO5212054 | FALSE | FALSE | TRUE | NCIt | 24.02d |
药物声明名称为BRAF(V600E) Kinase Inhibitor RO5212054,药物命名法类型是Primary Drug Name,药物概念 ID 是ncit:C92591,药物名称为BRAF(V600E) KINASE INHIBITOR RO5212054。该药物未被批准(approved为FALSE),不是免疫疗法(immunotherapy为FALSE),是抗肿瘤药物(anti_neoplastic为TRUE) 。药物数据的来源数据库名称是NCIt,数据库版本为24.02d。
表三:gene.tsv
其中的各列的意思分别是
- gene_claim_name:基因的声明名称。
- nomenclature:基因命名法类型。
- concept_id:基因的唯一标识代码。
- gene_name:基因的标准正式名。
- source_db_name:基因数据的来源数据库名。
- source_db_version:来源数据库的版本。
| gene_claim_name | nomenclature | concept_id | gene_name | source_db_name | source_db_version |
|---|---|---|---|---|---|
| NGFIBA | NCBI Gene Name | NULL | NULL | BaderLab | Feb-14 |
基因声明名称为NGFIBA,基因命名法类型是NCBI Gene Name,基因概念 ID 是NULL,基因名称同样为NULL。基因数据的来源数据库名称是BaderLab,数据库版本为Feb-14。
表四:categories.tsv
其中的各列的意思分别是
- name:某实体的名称(这里可能是基因或其他生物相关实体名称)。
- name-2:该实体的另一个相关名称或描述。
- source_db_name:数据的来源数据库名。
- source_db_version:来源数据库的版本。
| name | name-2 | source_db_name | source_db_version |
|---|---|---|---|
| PXR | NUCLEAR HORMONE RECEPTOR | BaderLab | Feb-14 |
名称为PXR,另一个相关名称或描述是NUCLEAR HORMONE RECEPTOR。数据的来源数据库名称是BaderLab,数据库版本为Feb-14。
DrugBank
- DrugBank
- 综合药物信息数据库,整合了药物的化学、药理、毒理等多方面信息。
- 一个巨大的数据库。这个数据库免费开放给学术用户,但商业使用收费。可以在网页上进行学生/老师认证注册。药物再利用在其中是很小的一个板块,可以在这里下载。
- 学生身份目前正在审核状态,无法下载。
ChEMBL
- ChEMBL
- ChEMBL 是一个开源的生物活性分子数据库,专注于小分子化合物与生物靶点之间的相互作用信息。
- 数据大小大约2GB。有不同的版本,提供FTP方式下载,可以在这里下载最新的版本ChEMBL_35。
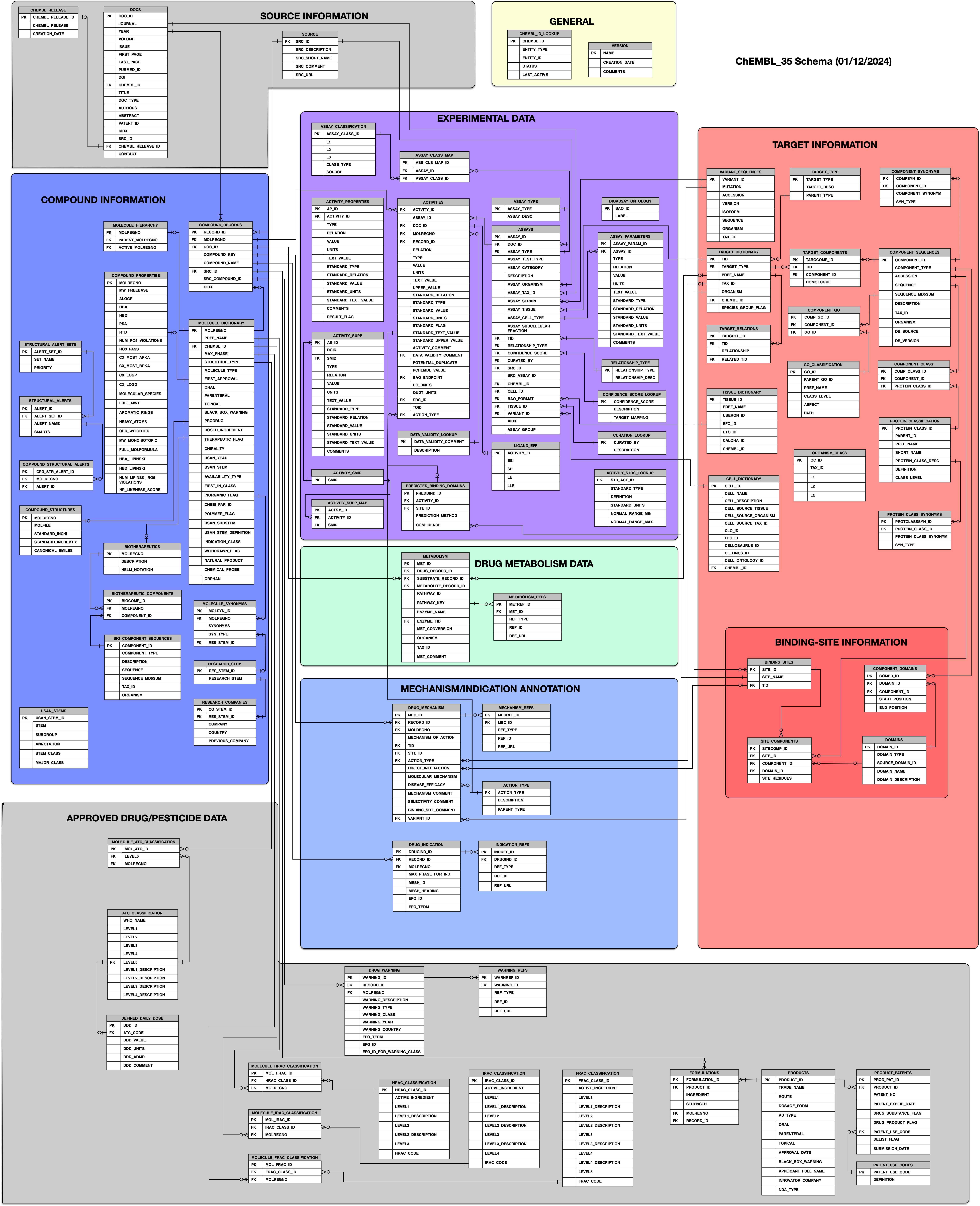
- 目录中的内容可以参考README文件。主要的部分可以见下图,组织时FASTA文件、SDF文件、HTML文件和TXT文件都有使用。数据可以加载到MySQL、PostgreSQL等数据库中。
- 有官方的数据库结构图如下,主要分成
- 化合物信息:主要以蓝色区域呈现,记录了化合物的结构、理化性质等,像分子式、分子量和二维结构这些
- 实验数据:用紫色区域表示,包含化合物与靶点相互作用的活性数据,还有实验条件,比如不同化合物对特定靶点的结合亲和力数值,以及实验采用的检测方法等
- 靶点和结合位点信息:红色区域代表这部分内容。靶点信息涉及蛋白质靶点的氨基酸序列、三维结构和功能分类;结合位点信息聚焦靶点与化合物结合的区域,包括氨基酸组成、空间结构和相互作用模式
- 药物代谢数据:浅绿色区域涵盖这部分,记录了药物在体内的代谢途径、产物和酶——我们可以通过这部分预测药物疗效、毒性以及药物之间的相互作用。
- 作用机制 / 药物注释:浅蓝色区域负责注释药物作用机制,关联化合物、靶点和生物效应。
- 来源和已批准药物数据:灰色区域中,来源信息保证数据可追溯,已批准药物 数据包含批准文号、适应症和生产厂家等,这个应该是用来避免专利壁垒的,可以查看专利即将到期的药物。
- 常规信息:浅黄色区域存放数据库的版本号、更新时间等基础信息。
BindingDB
- BindingDB
- 主要收集药物靶点蛋白质和类药小分子之间的相互作用亲和力数据。数据库大小为484.07 MB(TSV版本)。
- 该数据文件只有一个表格,该表总共包含117列。
- 配体标识与结构(7 列):如 “BindingDB MonomerID” 等,用于确定和描述配体,包括多种结构表示方式和自定义名称。
- 靶点基础信息(2 列):“Target Name” 和 “Target Source Organism According to Curator or DataSource”,明确靶点名称及所属生物。
- 结合活性数据(6 列):“Ki (nM)” 等,反映配体与靶点结合的强度和速率。
- 实验环境参数(2 列):“pH” 和 “Temp (°C)”,体现结合数据测量时的环境条件。
- 数据与文献来源(7 列):“Curation/DataSource” 及各类文献标识符,用于追溯数据出处和原始研究。
- 数据库交叉引用(14 列):含 “PubChem CID” 等多个数据库的配体标识,方便数据整合查询。
- 蛋白质链详情(80 列):先以 “Number of Protein Chains in Target” 记录链数量,后续多列针对每条链,涵盖序列、结构 ID、UniProt 相关名称和 ID 等信息。
表格中的信息以第一条记录(数据行 ID 为 1)为例,大概描述了:
- 配体相关信息:BindingDB MonomerID 为
608734,Ligand SMILES 为 “O [C@@H] 1C@@HC@@HN (CCCCCC (O)=O) C (=O) N (CCCCCC (O)=O)[C@@H] 1Cc1ccccc1”,其对应的 BindingDB Ligand Name 是 “6-[(4R,5S,6S,7R)-4,7 - 二苄基 - 3-(5 - 羧基戊基)-5,6 - 二羟基 - 2 - 氧代 - 1,3 - 二氮杂环庚烷 - 1 - 基] 己酸::DMPC 环脲 1”。- 靶点相关信息:Target Name 为 “Dimer of Gag-Pol polyprotein [501 - 599]”,表明靶点是 Gag-Pol 多聚蛋白二聚体的 501 - 599 区域;Target Source Organism 为 “Human immunodeficiency virus 1”,即靶点来源于人类免疫缺陷病毒 1。
- 结合活性数据:Ki (nM) 为 0.24 ,显示配体与靶点的结合亲和力较强。
- 实验条件:测量时 pH 为 5.5,温度为 37°C。
- 数据来源:由 BindingDB 从文献整理而来,相关文献的 DOI 是 10.1021/jm9602571,PMID 为 8784449,可据此追溯原始研究。
- 数据库交叉引用:PubChem CID 为 3009304,PubChem SID 为 483500124,方便在 PubChem 数据库中查找该配体更多信息。
- 蛋白质链信息:靶点含 1 条蛋白质链,其序列为 “PQITLWQRPLVTIKIGGQLKEALLDTGADDTVLEEMSLPGRWKPKMIGGIGGFIKVRQYDQILIEICGHKAIGTVLVGPTPVNIIGRNLLTQIGCTLNF”,相关 PDB ID 有 “1W5Y”“1W5X” 等多个,UniProt(SwissProt)相关信息显示,其推荐名称是 “Gag-Pol polyprotein”,Entry Name 为 “POL_HV1BR”,Primary ID 为 “P03367”。